Изучение метилирования сайтов RCGY в регуляторной области гена FAM19A4 в препаратах ДНК клеточных линий человека методом GLAD-ПЦР анализа
Акишев А.Г., Нетесова Н.А., Абдурашитов М.А., Дегтярев С.Х.
А.Г. Акишев1 *, Н.А. Нетесова2, М.А. Абдурашитов1, С.Х. Дегтярев1
1 НПО «СибЭнзим» Новосибирск
2 ООО «Эпигенлаб» Новосибирск
* автор для переписки: Александр Акишев, НПО СибЭнзим, ул. Тимакова, д. 2/12 , Новосибирск, 630117, Россия; тел.: +7(383)333-4991; факс: +7(383)333-6853; E-mail: aki@sibenzyme.ru
Методом GLAD-ПЦР анализа определяли метилирования сайтов RCGY в регуляторной области гена FAM19A4 (Хр3: 68932451 — 68932800) в препаратах ДНК малигнантных клеточных линий человека Raji, Jurkat, HeLa, U-937 и контрольной ДНК клеточной линии фибробластов легких L68.
Показано, что октануклеотид GCGCGCGC (Хр3: 68932500), находящийся в первом экзоне гена FAM19A4, расщепляется приблизительно в равной степени в ДНК всех 4-х малигнантных клеточных линий человека, тогда как в ДНК L68 этом сайт гидролизуется в 10-15 раз слабее. Эти результаты соответствуют полученным ранее данным эпигеномного секвенирования ДНК L68, Raji и U-937.
Метилирование CG-динуклеотидов в позициях 68932727 и/или 68932740 в районе промотора гена FAM19A4 происходит только в опухолевых ДНК и не наблюдается в ДНК L68, а метилирование CG-динуклеотида в позиции 68932704 наблюдается только в ДНК HeLa.
Ключевые слова: метилирование ДНК, ДНК из крови человека, GLAD-ПЦР анализ, ген FAM19A4
DOI: 10.26213/SE.2021.97.62.001
Данные для цитирования:
Акишев А.Г., Нетесова Н.А., Абдурашитов М.А., Дегтярев С.Х. (2021) Изучение метилирования сайтов RCGY в регуляторной области гена FAM19A4 в препаратах ДНК клеточных линий человека методом GLAD-ПЦР анализа, Эпигенетич ДНК диагност, том 2021(1), с.31-40, DOI: 10.26213/SE.2021.97.62.001

Эта статья доступна по лицензии Creative Commons Attribution-NonCommercial-NoDerivatives 4.0 International
Введение
Аберрантное метилирование сайтов RCGY в регуляторных участках генов-онкосупрессоров наблюдается на начальных стадиях развития злокачественных новообразований [1]. Исследование этой модификации необходимо для создания панели сайтов RCGY, являющихся эпигенетическими маркерами для диагностики онкологических заболеваний на ранних стадиях.
Ранее мы изучали аберрантное метилирование регуляторных областей целого ряда генов в ДНК опухолевых тканей в случае колоректального рака [2], рака желудка [3] и рака легких [4] методом GLAD-ПЦР анализа, который был разработан нами ранее [5]. Проведение GLAD-ПЦР анализа включает три стадии [2]. На первой стадии изучаемая ДНК расщепляется MD ДНК-эндонуклеазой GlaI, на второй стадии к полученным фрагментам подшивается уникальный адаптер и на 3-ей стадии проводится ПЦР в реальном времени с праймера, комплементарного изучаемому фрагменту ДНК, и второго (так называемого гибридного) праймера, комплементарного уникальному адаптеру и следующим нескольким нуклеотидам геномной последовательности в точке расщепления целевого сайта. В результате при наличии огромного количества разных фрагментов, полученных после гидролиза ДНК и последующей сшивки, ПЦР идет преимущественно с того фрагмента, который расположен рядом с целевым сайтом R(5mC)GY. Использование зонда, также имеющего уникальную структуру, позволяет еще больше повысить специфичность реакции.
В данной работе методом GLAD-ПЦР анализа мы провели определение метилирования ряда сайтов RCGY в регуляторной области гена FAM19A4 в препаратах ДНК 4-х малигнантных клеточных линий человека и контрольной ДНК клеточной линии фибробластов легких L68. Данный ген кодирует небольшой секретируемый хемокин-подобный белок с неустановленной функцией и в нескольких публикациях рассматривается как перспективный эпимаркер для диагностики рака шейки матки [14-16].
Материалы и методы
Для проведения экспериментов использовали набор для определения статуса метилирования сайтов RCGY в регуляторной области гена FAM19A4 методом GLAD-ПЦР анализа (ООО «СибЭнзайм», кат. номер K013S) согласно прилагаемой производителем инструкции по применению. В работе использовались ДНК клеточной линии L68 (фибробласты легких) и малигнантных клеточных линий Raji (лимфома Беркитта), U-937 (гистиоцитарная лимфома), Jurkat (Т-лимфобластная лейкемия), HeLa (аденокарцинома шейки матки).
Изучаемый фрагмент ДНК был разбит на 2 участка, для каждого из которых проводился ПЦР с пары геномной праймер – зонд, структура которых была выбрана для данного участка, и набора гибридных праймеров. Для изучения метилирования сайтов RCGY в участке ДНК 68932451 – 68932536 были использованы геномный праймер, зонд и гибридные праймеры следующей структуры
FAM19A4_618 5′ GGAGAAGCGGCAGCTGAGGC 3′ (20)
FAM19A4z641 5′ FAM-ACTCCCGATGTTTGCCCGCAGCC-BHQ1 3′ (23)
HP11 5′ CCTGCTCTTTCATCGGCGG 3′(19) — 1-ый сайт
HP10 5′ CCTGCTCTTTCATCGGCGC 3′(19) — 2-ой, 3-ий и 6-ой сайты
HP2 5′ CCTGCTCTTTCATCGGCAC 3′(19 – 4-ой сайт
HP7 5′ CCTGCTCTTTCATCGGCCG 3′(19) – 5-ый сайт
ПЦР в реальном времени проводили согласно протоколу производителя [N] в объеме 20 мкл на детектирующем амплификаторе CFX-96 («Bio-Rad», США) по следующей программе:
- при 95°C — 3 мин, при 96°C — 30 сек;
- 5 «слепых» циклов: 96°C — 10 сек, 64°C — 20 сек, 72°C — 10 сек, 75°C — 5 сек;
- 40 циклов: 96°C — 10 сек, 64°C — 20 сек (с детекцией флуоресцентного сигнала в канале FAM), 72°C — 10 сек, 75°C — 5 сек.
Для изучения метилирования сайтов RCGY в участке ДНК 68932700 — 68932789были использованы геномный праймер, зонд и гибридные праймеры следующей структуры
FAM1.5z 5′ FAM-CGC TGT GGC CAC CAC TCC GC-BHQ1 3′ (20)
FAM1.5r 5′ GGA AGA ATA CCT TTT GCT CAG CAC CT 3′ (26)
HP8: 5′ CCTGCTCTTTCATCGGCCT 3′- 1-ый и 2—ой сайты
HP6: 5′ CCTGCTCTTTCATCGGCCC 3′- 3-ий сайт
HP10 5′ CCTGCTCTTTCATCGGCGC 3′(19) 4-ый сайт
HP27 5′ CCTGCTCTTTCATCGGTGG 3′(19) 5-ый сайт
ПЦР в реальном времени проводили согласно протоколу производителя [6] в объеме 20 мкл на детектирующем амплификаторе CFX-96 («Bio-Rad», США) по следующей программе:
- при 95°C — 3 мин, при 96°C — 30 сек;
- 5 «слепых» циклов: 95°C — 10 сек, 58°C — 20 сек, 72°C — 10 сек, 75°C — 5 сек;
- 40 циклов: 95°C — 10 сек, 58°C — 20 сек (с детекцией флуоресцентного сигнала в канале FAM), 72°C — 10 сек, 75°C — 5 сек.
Результаты и обсуждение
Для выявления модифицированных CG-динуклеотидов в геномной ДНК человека используют технологию бисульфитной обработки ДНК и последующего определения нуклеотидной последовательности полученного продукта [7]. На сегодня широко применение нашли ДНК-чипы Illumina Infinium 450k, которые позволяет выявить метилированные CG-динуклеотиды в более чем 450000 позициях генома человека. Ранее мы разработали метод эпигеномного секвенирования геномной ДНК человека на NGS машине, позволяющий выявить более 2 млн сайтов RCGY, метилирование которых наблюдается в ДНК клеточных линий человека [8]. Методом эпигеномного секвенирования на машине Illumina Genome Analyzer IIx мы определяли локализацию сайтов R(5mC)GY в геномной ДНК малигнантных клеточных линий Raji (лимфома Беркитта), U-937 (гистиоцитарная лимфома) и контрольной ДНК L68 [9]. Полученные данные позволяют сравнить метилирование сайтов RCGY в регуляторных участках различных генов в ДНК этих трех клеточных линий человека.
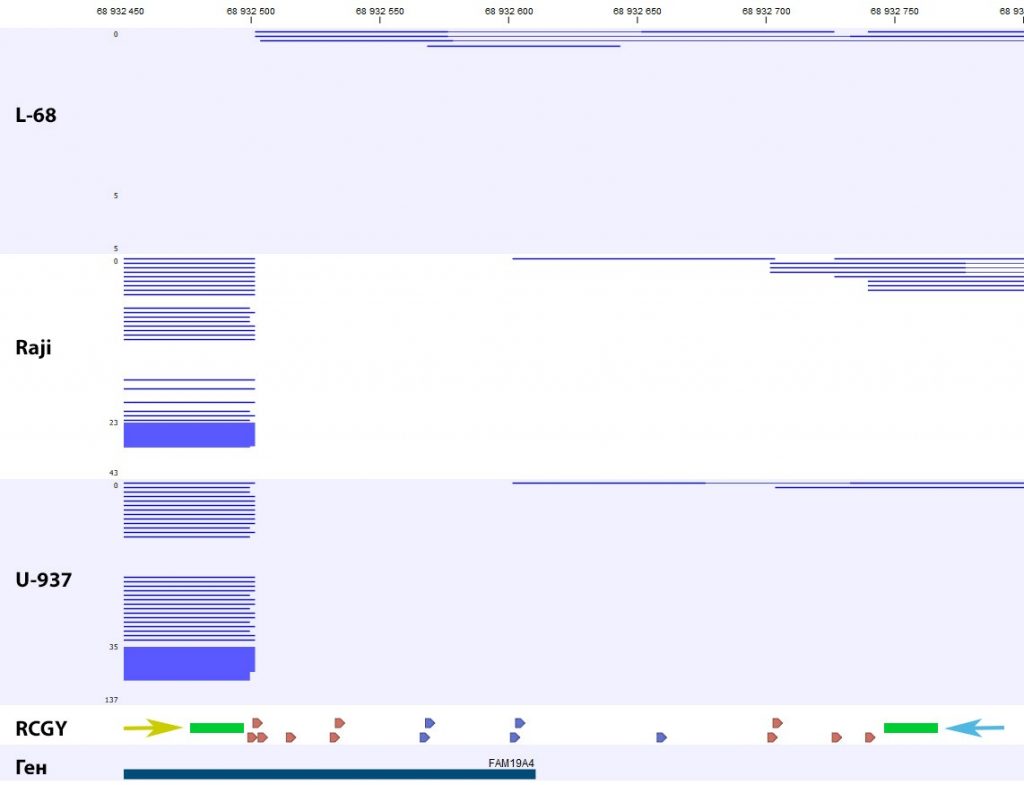
В частности, на рисунке 1 представлена нуклеотидная последовательность фрагмента ДНК третьей хромосомы человека (Хр3: 68932451 — 68932800), являющаяся частью регуляторной области гена FAM19A4.
Рис. 1. Нуклеотидная последовательность регуляторной области гена FAM19A4.
Районы связывания геномных праймеров выделены желтым и синим фоном, а зона связывания флюоресцентно-меченых зондов выделена зеленым цветом. Последовательности RCGY, являющиеся сайтом метилирования ДНК-метилтрансферазой DNMT3 [12] и, в случае их модификации, субстратом MD ДНК-эндонуклеазы GlaI [13], подчеркнуты двойной линией. Сайты RCGY, ближайшие к парам праймер-зонд, обозначены шрифтом красного цвета. CG-динуклеотиды, проверяемые с помощью чипа Illumina Infinium 450k и находящиеся в данном фрагменте ДНК, выделены серым фоном (последовательно cg12417685, cg12219082, cg12412079, cg13921352). Лиловым цветом выделен начальный нуклеотид гена по системе GenCode.
На рисунке 2 представлены результаты определения сайтов R(5mC)GY в изучаемом фрагменте ДНК методом NGS на машине Illumina Genome Analyzer IIx [10]. В нижней части рисунка 2 треугольниками отмечено положение сайтов RCGY, которое соответствует позициям этих сайтов на рисунке 1. Концы фрагментов ДНК на рисунке 2 соответствуют позициям расщеплений и, следовательно, присутствию метилированного CG-динуклеотида в составе сайта RCGY. Как видно из рисунка обе ДНК из малигнантных клеточных линий расщепляются в октануклеотиде GCGCGCGC, находящимся в первом экзоне в положении 68932500, тогда как существенного гидролиза ДНК L68 в этом положении не наблюдается.
В изучаемом фрагменте ДНК в позициях 68932702, 68932704, 68932727 и 68932740 находятся 4 CG-динуклеотида под номерами cg12417685, cg12219082, cg12412079, cg13921352, соответственно, проверяемые ДНК-чипом Illumina Infinium 450k в этой области генома. Данные «The Cancer Genome Atlas» (TCGA) показывают, что данные динуклеотиды часто метилированы в опухолевых клетках при раке шейки матки [11], однако в случае ДНК клеточных линий L68, Raji и U-937 в NGS-данных не наблюдается существенной разницы в модификации данных CG-динуклеотидов.
В представленной на рисунке 1 нуклеотидной последовательности нами были выбраны 2 участка ДНК (1 в промоторной области и 1 в зоне первого экзона), в которых были размещены праймеры и зонды для ПЦР. Сайты RCGY, ближайшие к парам праймер-зонд, обозначены шрифтом красного цвета. В случае второй пары праймер-зонд красным шрифтом обозначены два сайта поскольку они оба имеют структуру GCGC и у них совпадает динуклеотид CT, прилегающий к сайту со стороны зонда. Эти сайты будут амплифицироваться с одного и того же гибридного праймера.
В первом участке, в зоне первого экзона, GLAD-ПЦР анализ позволяет определить метилирование первого CG-динуклеотида в сайте GCGC (обозначенный красным шрифтом) в положении 68932500 в первом экзоне при использовании гибридного праймера HP11 5′ CCTGCTCTTTCATCGGCGG 3′. При неметилированном (или частично метилированным) первом CG-динуклеотиде возможно определение метилирования двух остальных CG-динуклеотидов в этом сайте (одного из них или их обоих) при использовании гибридного праймера HP10 5′ CCTGCTCTTTCATCGGCGC 3′. На расстоянии 7 нуклеотидов от данного октануклеотида находится сайт GCGC, метилирование которого (в случае отсутствия или частичной модификации цитозинов в октануклеотиде) может быть установлено с помощью гибридного праймера HP2 5′ CCTGCTCTTTCATCGGCAC 3′. И, наконец, еще дальше, на расстоянии 13 нуклеотидов находится сайт GCGCGC, метилирование которого может быть выявлено при отсутствии модификации предыдущих сайтов и в котором метилирование первого CG-динуклеотида может быть установлено с помощью гибридного праймера HP7 5′ CCTGCTCTTTCATCGGCCG 3′, а метилирование второго CG-динуклеотида (в случае отсутствия еще и модификации первого CG-динуклеотида) определяется гибридным праймером HP10 5′ CCTGCTCTTTCATCGGCGC 3′.
Во втором участке, в промоторной области гена первые два сайта, прилегающие к области связывания зонда, определяются GLAD-ПЦР анализом при использовании гибридного праймера HP8 5′ CCTGCTCTTTCATCGGCCT 3′. С помощью гибридного праймера HP6 5′ CCTGCTCTTTCATCGGCCC 3′ определяется метилирование второго CG-динуклеотида в сайте GCGCGC в положении 68932704 (при отсутствии или частичном метилировании первых двух сайтов). И, наконец, метилирование первого CG-динуклеотида в сайте GCGCGC в положении 68932702 (при отсутствии модификации или частичном метилировании предыдущих сайтов) определяется при использовании гибридного праймера HP10 5′ CCTGCTCTTTCATCGGCGC 3′.
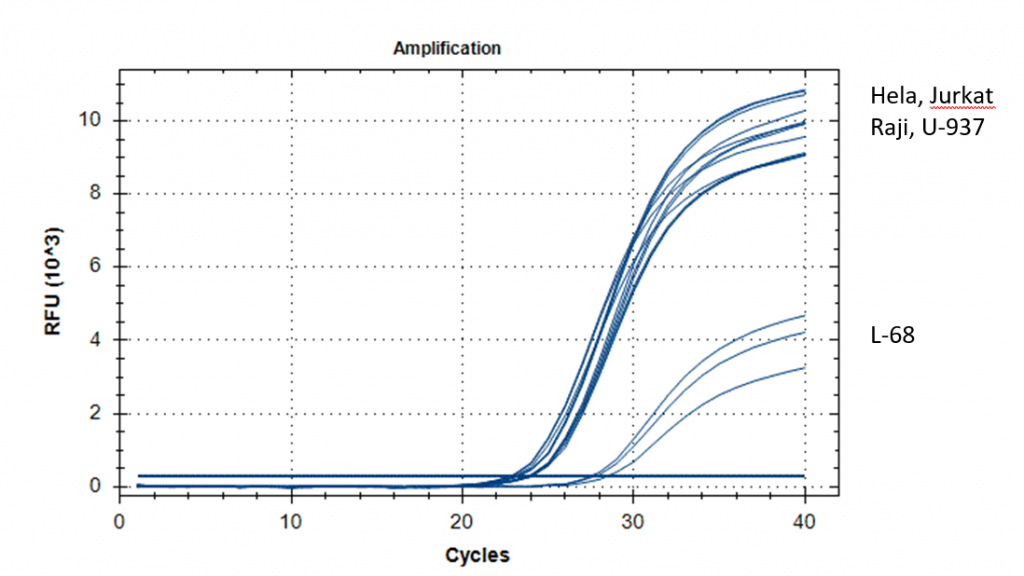
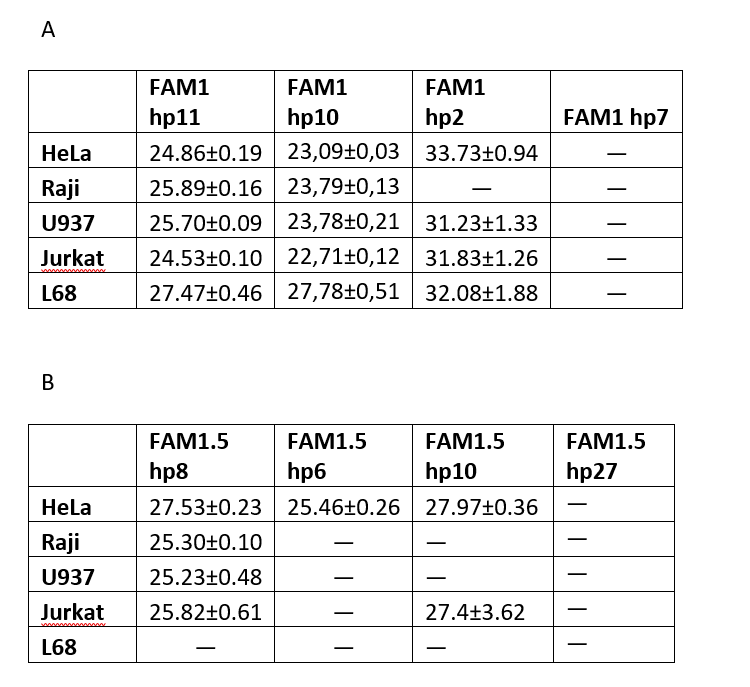
На рисунке 3 представлены кривые роста флуоресценции в ходе GLAD-ПЦР анализа первого участка ДНК с использованием гибридного праймера HP10, а в таблице 1A приведены величины Cq, полученные в ходе GLAD-ПЦР анализа для этого и всех остальных гибридных праймеров по данному участку ДНК. Как видно из рис.3 и таблицы 1A при использовании гибридного праймера HP10 для ДНК всех малигнантных клеточных линий наблюдается достаточно высокий уровень метилирования (Cq 22,7-23,8), тогда как ДНК L68 дает Cq только 27,8. При этом в ДНК малигнантных клеточных линий уровень метилирования 2-го и/или 3-го CG-динуклеотида в сайте GCGCGCGC в положениях 68932502 и 68932504 в 3-4 раза выше, чем первого CG-динуклеотида в положении 68932500. В то же время в ДНК L68 уровень метилирования первого и остальных динуклеотидов существенно меньше, чем в опухолевых ДНК, и между собой отличается незначительно. Согласно данным таблицы 1A уровень метилирования сайта GCGC, определяемого с гибридным праймером HP2, близок к фоновым значениям (Cq более 31), а в случае гибридного праймера HP7 GLAD-ПЦР анализ определяет анализируемый сайт как неметилированный.
Таким образом, октануклеотид GCGCGCGC (Хр3: 68932500), находящийся в первом экзоне гена FAM19A4, расщепляется приблизительно в равной степени в ДНК всех 4-х малигнантных клеточных линий человека, тогда как в ДНК L68 этом сайт гидролизуется в 10-15 раз слабее. Эти результаты соответствуют полученным ранее данным эпигеномного секвенирования ДНК L68, Raji и U-937.
В таблице 1B представлены результаты GLAD-ПЦР анализа второго участка ДНК. Как видно из таблицы метилирование изучаемых сайтов во втором участке ДНК наблюдается только в малигнантных клеточных линиях и не происходит в ДНК L68. При этом метилирование двух сайтов, ближайших к участку связывания зонда, происходит в трех ДНК опухолевых клеточных линий приблизительно на одном уровне (Cq = 25,23 – 25,82), тогда как уровень метилирования этих сайтов в ДНК HeLa меньше (Cq = 27,53). В то же время метилирование СG-динуклеотида в позиции 68932704 (третий сайт от зоны связывания зонда) наблюдается только в ДНК HeLa и его уровень (Cq = 25,46) совпадает с уровнем метилирования первых двух сайтов в остальных малигнантных ДНК. Таким образом, мы наблюдаем метилирование 4-х CG-динуклеотидов в позициях 68932702, 68932704, 68932727 и 68932740 только в опухолевых ДНК, при этом метилирование в позиции 68932704 наблюдается только в ДНК HeLa. Полученные данные согласуются с результатами, представленными в «The Cancer Genome Atlas» (TCGA), в которых показано метилирование этих 4-х CG-динуклеотидов в опухолевой ДНК при раке шейки матки [11].
Таблица 1
Значения Cq в GLAD-ПЦР анализе ДНК пяти клеточных линий с использованием различных сочетаний зонда и гибридных праймеров.
Литература
- Kulis M, Esteller M. DNA methylation and cancer. Adv Genet. 2010;70:27-56.
- Evdokimov A.A., Nina A. Netesova N.A., Smetannikova N.A. et al. GLAD-PCR Assay of DNA Methylation Markers Associated with Colorectal Cancer // Biol Med (Aligarh) , 8:7(2016).
- Б. С. Малышев, Н. А. Нетесова, Н. А. Сметанникова, М. А. Абдурашитов, А. Г. Акишев, Е. В. Дубинин, А. З. Азанов, И. В. Вихлянов, М. К. Никитин, А. Б. Карпов, С. Х. Дегтярев GlaD-ПЦР-анализ сайтов метилирования ДНК в регуляторных областях генов-онкосупрессоров при раке желудка//Acta Naturae, 2020, том 12, № 3(46): 124-133.
- Smetannikova NA, Evdokimov AA, Netesova NA, Abdurashitov MA, Akishev AG, Dubinin EV, Pozdnyakov PI, Vihlyanov IV, Nikitin MK, Topolnitsky EB, Karpov AB, Kolomiets SA, Degtyarev SK Application of GLAD-PCR Assay for Study on DNA Methylation in Regulatory Regions of Some Tumor-Suppressor Genes in Lung Cancer// Zhongguo Fei Ai Za Zhi. 2019;22(9):551-561.
- Гончар Д.А., Акишев А.Г., Дегтярев С.Х. BlsI- и GlaI- ПЦР анализ – новый метод исследования метилированных участков ДНК. // Вестник биотехнологии и физико-химической биологии имени Ю. А. Овчинникова, Т. 6 , № 1 ,с. 5-12 (2010).
- Системы ПЦР реального времени с флуоресцентной детекцией. Руководство по эксплуатации. https://www.bio-rad.com/webroot/web/pdf/lsr/russian_federation/russian/literature/10021337_ru.pdf
- Kurdyukov S, Bullock M. DNA Methylation Analysis: Choosing the Right Method. Biology (Basel). 2016;5(1):3.
- Abdurashitov M.A., Tomilov V.N., Gonchar D.A., Kuznetsov V.V., Degtyarev S.Kh. Mapping of R(5mC)GY Sites in the Genome of Human Malignant Cell Line Raji // Biol Med (Aligarh) 7(4), BM-135-15 (2015).
- Murat A. Abdurashitov, Viktor N. Tomilov, Danila A. Gonchar, Anastasiya V. Snezhkina, George S. Krasnov, Anna V. Kudryavtseva and Sergey Kh. Degtyarev Comparative analysis of RCGY sites methylation in three human cell lines// Epigenetic DNA diagnostics, (2019).
- Specification Sheet: Genome AnalyzerIIx System https://support.illumina.com/content/dam/illumina-marketing/documents/products/datasheets/datasheet_genome_analyzeriix.pdf
- The Cancer Genome Atlas Program https://www.cancer.gov/tcga
- Handa V., Jeltsch A. Profound sequence preference of Dnmt3a and Dnmt3b mammalian DNA methyltransferases shape the human epigenome // J. Mol. Biol. – 2005. – Vol. 348. – P. 1103–1112.
- Tarasova G.V., Nayakshina T.N., Degtyarev S.K. Substrate specificity of new methyl-directed DNA endonuclease GlaI // BMC Mol. Biol. – 2008. – Vol. 9. – N. 7.
- De Strooper LM, Meijer CJ, Berkhof J, Hesselink AT, Snijders PJ, Steenbergen RD, Heideman DA. Methylation analysis of the FAM19A4 gene in cervical scrapes is highly efficient in detecting cervical carcinomas and advanced CIN2/3 lesions. Cancer Prev Res (Phila). 2014;7(12):1251-1257.
- De Strooper LMA, Verhoef VMJ, Berkhof J, Hesselink AT, de Bruin HME, van Kemenade FJ, Bosgraaf RP, Bekkers RLM, Massuger LFAG, Melchers WJG, Steenbergen RDM, Snijders PJF, Meijer CJLM, Heideman DAM. Validation of the FAM19A4/mir124-2 DNA methylation test for both lavage- and brush-based self-samples to detect cervical (pre)cancer in HPV-positive women. Gynecol Oncol. 2016;141(2):341-347.
- Wang SF, Bu QW, Zhang L, Ma JW, Heng Y, Luo JQ, Luo XP. Detection and analysis of FAM19A4 promoter methylation in cervical exfoliated cells. Zhonghua Yi Xue Za Zhi. 2019;99(25):1963-1967.