Определение полиморфизма 5mC/T в повторе AluSx (CHR16: 75033884) в препаратах ДНК из крови человека методами GlaI- и FatI-ПЦР анализа
А.Г. Акишев1 *, Н.А. Нетесова2, М.А. Абдурашитов1, С.Х. Дегтярев1
1 НПО «СибЭнзим» Новосибирск
2 ООО «Эпигенлаб» Новосибирск
* автор для переписки: Александр Акишев, НПО СибЭнзим, ул. Тимакова, д. 2/12 , Новосибирск, 630117, Россия; тел.: +7(383)333-4991; факс: +7(383)333-6853; E-mail: aki@sibenzyme.ru
Методами GlaI- и FatI-ПЦР анализа определяли частоты вариантов полиморфизма 5mC/T в повторе AluSx в положении хр16: 75033884 (по геномной сборке GRCh38.p13) в препаратах ДНК, выделенной из клеток крови 92 человек. Исследование включало (1) выделение лейкоцитарной ДНК из клеток крови, (2) проведение GlaI- и FatI-ПЦР анализа фрагмента ДНК хр16: 75033860 — 75033957, (3) определение 5-метилцитозина и тимина в позиции (хр16: 75033884) в анализируемых препаратах ДНК и (4) сравнительный анализ полученных результатов.
Показано, что 53 донора (57,61%) имеют гетерозиготный набор C/T в положении хр16: 75033884, 19 доноров (20,65%) гомозиготны по T, а 20 доноров (21,74%) – по C. Таким образом, принимая во внимание, что клетки крови имеют диплоидный набор хромосом, замена C на T встречается в 91 из 184 проанализированных вариантов (49,46%). При этом из полученных результатов следует, что цитозин в положении хр16: 75033884 в бОльшей части молекул ДНК находится в метилированной форме (5-метилцитозин), что согласуется с положением о преимущественном метилировании CG-динуклеотидов в Alu-повторах.
Ключевые слова: однонуклеотидный полиморфизм 5mC/T, ДНК из крови человека, GlaI-ПЦР анализ, FatI-ПЦР анализ
DOI: 10.26213/SE.2021.11.76.001
Данные для цитирования:
А.Г. Акишев, Н.А. Нетесова, М.А. Абдурашитов, Дегтярев (2021) Определение полиморфизма 5mC/T в повторе AluSx (CHR16: 75033884) в препаратах ДНК из крови человека методами GlaI- и FatI-ПЦР анализа, Эпигенетич ДНК диагност, том 2021(1), DOI: 10.26213/SE.2021.11.76.001

Эта статья доступна по лицензии Creative Commons Attribution-NonCommercial-NoDerivatives 4.0 International
Введение
Работа по определению генома человека, проводимая в рамках многочисленных научных программ, позволила установить последовательности ДНК множества людей и выявить в них те основания, по которым наблюдается полиморфизм. Однонуклеотидный полиморфизм (SNP) чаще всего представляет собой замену цитозина на тимин или, если смотреть по комплементарной цепи, гуанина на аденин. Данное изменение обычно происходит вследствие дезаминирования 5-метилцитозина и превращения его в тимин и отмечается в самых различных элементах генома, включая и Alu-повторы [1]. В частности, было обнаружено наличие однонуклеотидного полиморфизма C/T в положении Chr16: 75033884 (rs66499664 по базе данных dbSNP), расположенного в повторе AluSx [2]. Данный повтор, в свою очередь, расположен в интроне гена ZNRF1, кодирующего убиквитинлигазу E3. Определение последовательности ДНК людей с использованием NGS-машин и последующее сравнение структуры различных геномов позволяет установить наличие полиморфизма и частоты вариантов в том или ином положении ДНК. Однако такое исследование не позволяет установить статус метилирования искомого цитозина, а также процент гомо- и гетерозигот по вариантам SNP.
В данной работе мы определяли полиморфизм 5mC/T в положении хр16: 75033884 (по геномной сборке GRCh38) в препаратах ДНК 92 доноров. На рисунке 1 представлен фрагмент ДНК хр16: 75033786-75033985. Подчеркиванием на рисунке обозначен повтор AluSx, внутри которого находится сайт ACGC (обозначен двойным подчеркиванием), содержащий, в свою очередь, искомый остаток цитозина (выделен красным цветом). Как видно из структуры указанного фрагмента, при наличии в данном сайте метилированного CG-динуклеотида в обеих цепях ДНК, образуется последовательность 5′-A(5mC)GC-3’/3′-TG(5mC)G-5′, которая является сайтом узнавания MD-эндонуклеазы GlaI [3]. В тоже время, в случае замены основания происходит превращение искомого цитозина в тимин с образованием последовательности (в положении хр16: 75033882 – 75033885) CATG, являющейся сайтом узнавания рестриктазы FatI.
На рисунке 1 представлена структура фрагмента ДНК хр16: 75033786-75033985 и указаны зоны связывания праймеров (выделены желтым и синим) и зонда (выделенного зеленым), используемых в ПЦР. Нахождение одного из праймеров вне структуры повтора AluSx приводит к проведению ПЦР только с данного фрагмента ДНК.
Рисунок 1.
Нуклеотидная последовательность анализируемого участка геномной ДНК

Методы FatI- и GlaI-ПЦР анализа основаны на расщеплении ДНК ферментами FatI или GlaI, соответственно, с последующей ПЦР в реальном времени с использованием зонда и праймеров, расположенных вокруг анализируемого участка ДНК. При этом ферменты FatI и GlaI не имеют других сайтов узнавания в данном фрагменте ДНК. В нашей работе мы использовали праймеры и зонд, структура которых указана в разделе Материалы и методы, а их положение в ДНК обозначено на рис.1. При наличии замены C на T ДНК расщепляется ферментом FatI и ПЦР не происходит (или дает фоновый сигнал в случае неполного гидролиза ДНК). Известно, что подавляющее большинство CG-динуклеотидов (96-99%) метилировано в Alu-повторах немалигнантных клеток [4], в этом случае в положении хр16: 75033884 находится 5-метилцитозин, входящий в состав сайта A(5mC)GC. Фермент GlaI расщепляет данный метилированный сайт, вследствие чего ПЦР не происходит или дает фоновый сигнал в случае неполного гидролиза ДНК. В качестве контроля использовалась ДНК, расщепленная ферментом HinfI, сайты узнавания которого обозначены на рисунке фиолетовым цветом, и также проводился ПЦР в реальном времени. При использовании в качестве матрицы ДНК, гидролизованной HinfI, ПЦР дает значения Cq, являющиеся контрольной величиной для данного фрагмента, а при проведении ПЦР на ДНК, расщепленной GlaI или FatI, происходит увеличение Cq, соответствующее количеству разрезанного субстрата.
Материалы и методы
Материалы. В работе использовали фенол (ОАО «Реактив», Новосибирск), хлороформ (ОАО «Реактив», Новосибирск), изоамиловый спирт (ОАО «Реактив», Новосибирск), РНКазу А (ООО «Самсон-Мед», Санкт-Петербург), SDS (ООО «Хеликон», Москва). Набор для проведения GlaI-ПЦР анализа, набор для очистки ДНК из легкой фракции клеток крови, ДНК клеточных линий L68, HeLa, Raji и U-937, дезоксиолигонуклеотиды, ДНК фага лямбда, рестриктазы, HinfI, FatI, бидистиллированная вода и буферные растворы производства НПО «СибЭнзим». Флуоресцентно-меченный зонд производства компании «Синтол» (Москва).
Материалом для исследования служили образцы ДНК, выделенные из клеток крови 92 условно здоровых доноров. Выделение ДНК из крови большинства доноров проводили фенол-хлороформным методом как описано ранее [5] с некоторыми модификациями. В ходе очистки полученные препараты ДНК обрабатывались только РНКазой А без добавления рестриктазы TaqI. В случае образцов №40, 55, 56, 121, 173, 178, 205, 237, 240, 561, 571, 584, 587, 603 и 604 выделение ДНК проводили из легкой фракции клеток крови как описано ранее [6]. Концентрацию полученных препаратов ДНК определяли спектрофотометрически.
Гидролиз ДНК лейкоцитов МД ДНК-эндонуклеазой и рестриктазами HinfI и FatI проводили как описано в инструкции к набору для GlaI-ПЦР анализа в пробирках для ПЦР (200 мкл) по 3 шт. на каждый анализируемый образец. Дополнительно к набору использовали эндонуклеазы рестрикции HinfI и FatI.
- В каждой пробирке смешивали 13,5 мкл H2O и 1,5 мкл ДНК образца, доведенного до концентрации 18 нг/мкл. Таким образом, каждый препарат ДНК был представлен тремя пробирками под номерами N1, N2 и N3.
- В пробирке на 1,5 мл готовили общую реакционную смесь A на количество пробирок, равное 3N (где N – число анализируемых образцов ДНК), содержащую из расчета на одну пробирку: 12.27 мкл H2O + 3.15мкл 10X SE TMN буфера + 0.32 мкл БСА(10 мг/мл) + 0.04 мкл ДНК фага лямбда(18 нг/мкл) + 0.17 мкл β-меркаптоэтанола (200 мМ).
- Полученную реакционную смесь разливали на три равные части в 3 пробирки на 1,5 мл. В первую пробирку вносили HinfI, во вторую – FatI по 0.12 мкл, в третью добавляли МД ДНК-эндонуклеазу из набора для GlaI-ПЦР анализа из расчета 0,19 мкл на каждый анализируемый образец ДНК.
- Далее в пробирки N1 добавляли по 15.2 мкл реакционной смеси с HinfI, в пробирки N2 добавляли по 15.2 мкл реакционной смеси с FatI и в пробирки N3 — по 15.2 мкл реакционной смеси с МД ДНК-эндонуклеазой.
- Гидролиз ДНК проводили 40 минут при 37°С для пробирок N1 и N3, и при 55°С для пробирок N2.
- После инкубации сбрасывали капли на микроцентрифуге и добавляли по 30,2 мкл реакционной смеси B, содержащей (из расчета на 1 образец) воды 20.8 мкл, буфер GLADх10 6.3 мкл, БСА (10 мг/мл) 0.6 мкл, dNTP (10 мМ каждый) 1.2 мкл, смесь праймеров и зонда (10 µM каждый) 2.4 мкл и SP-Taq ДНК-полимеразу (5 ед/мкл) 0.4 мкл.
- После перемешивания из каждой пробирки ее содержимое (60 мкл) разносили по 20 мкл в лунку ПЦР-плашки на 96 лунок.
Для расчета специфических праймеров и зондов использовали нуклеотидные последовательности из базы данных GenBank (http://ncbi.nlm.nih.gov/genbank) по версии генома человека GRCh38/hg38, семейство программ “Vector NTI 11.5” (Invitrogen, США) и онлайн-ресурс «BLAST» (http://blast.ncbi.nlm.nih.gov). Ниже указана структура праймеров и флуоресцентно меченого зонда, используемых в работе:
16-883d0 5′ TGACAAAATACACTTAGCATAAAAATTACCC 3′(31)
16-883r0 5′ GCCCAGACTGGTCTCAAACTCCT 3′(23)
16-883z 5′ FAM-CAATCTGCCCACCTTAGCCTCCCAA-BHQ1 3′(25)
ПЦР в реальном времени проводили согласно протоколу производителя [7] в объеме 20 мкл на детектирующем амплификаторе CXF-96 («Bio-Rad», США) по следующей программе:
при 95°C — 3 мин;
5 «слепых» циклов: 95°C — 10 сек, 65°C — 20 сек, 72°C — 10 сек, 75°C — 5 сек;
40 циклов: 95°C — 10 сек, 65°C — 20 сек (с детекцией флуоресцентного сигнала в канале FAM), 72°C — 10 сек, 75°C — 5 сек.
По завершении ПЦР, при помощи программного обеспечения амплификатора “Bio-Rad CFX Manager v.2.1”, устанавливали среднее значение Cq и значение среднеквадратичного отклонения для анализируемых образцов N1, N2 и N3 и полученные данные заносили в таблицу.
Результаты и обсуждение
В работе изучались препараты ДНК лейкоцитов крови 92 доноров, полученные как стандартным методом фенольной очистки, так и с помощью набора для выделения ДНК [8]. В числе доноров были 46 мужчин и 46 женщин. В таблице 1 представлены пол и возраст доноров (столбики 2 и 3, соответственно), а также концентрация полученных препаратов ДНК (столбик 4) и характеристика их чистоты, определяемой отношением оптической плотности при 260 нм и 280 нм (столбик 5).
Таблица 1.
Пол и возраст доноров крови, концентрация и фотометрическая чистота полученной лейкоцитарной ДНК
| N донора | Пол | Возраст, лет | Концентрация ДНК, нг/мкл | А260/А280 | N донора | Пол | Возраст, лет | Концентрация ДНК, нг/мкл | А260/А280 | |
|---|---|---|---|---|---|---|---|---|---|---|
| 1172 | жен | 14 | 376 | 1.60 | 1111 | жен | 50 | 141 | 1.70 | |
| 1164 | жен | 17 | 252 | 1.70 | 1151 | жен | 50 | 225 | 1.70 | |
| 1147 | муж | 18 | 180 | 2.00 | 1087 | жен | 51 | 95 | 1.70 | |
| 1133 | муж | 18 | 55 | 1.70 | 1093 | муж | 51 | 201 | 1.70 | |
| 1187 | муж | 20 | 304 | 1.80 | 1129 | жен | 51 | 72 | 1.70 | |
| 1502 | жен | 20 | 461 | 1.70 | 1165 | муж | 51 | 356 | 1.60 | |
| 1487 | жен | 22 | 406 | 1.70 | 584 | муж | 53 | 72 | 1.90 | |
| 1304 | жен | 23 | 129 | 1.70 | 1159 | муж | 53 | 358 | 1.70 | |
| 1218 | муж | 24 | 317 | 1.70 | 1358 | муж | 53 | 784 | 1.80 | |
| 1379 | жен | 24 | 241 | 1.80 | 587 | жен | 54 | 41 | 1.90 | |
| 1243 | муж | 24 | 280 | 1.70 | 561 | муж | 56 | 50 | 1.80 | |
| 1332 | муж | 25 | 507 | 1.90 | 1280 | муж | 56 | 436 | 1.80 | |
| 1173 | жен | 27 | 232 | 1.70 | 1127 | жен | 57 | 123 | 1.70 | |
| 1200 | муж | 27 | 271 | 1.60 | 1135 | жен | 57 | 127 | 1.60 | |
| 1203 | муж | 27 | 309 | 1.60 | 1163 | жен | 57 | 157 | 1.70 | |
| 1209 | муж | 27 | 204 | 1.70 | 1130 | жен | 58 | 59 | 1.70 | |
| 1214 | жен | 27 | 265 | 1.60 | 1137 | жен | 59 | 299 | 1.60 | |
| 1329 | муж | 27 | 488 | 1.80 | 1348 | муж | 60 | 761 | 1.70 | |
| 1225 | муж | 28 | 114 | 1.90 | 1349 | муж | 62 | 355 | 1.80 | |
| 1226 | жен | 30 | 297 | 1.90 | 1294 | муж | 63 | 173 | 1.60 | |
| 1221 | жен | 31 | 263 | 1.70 | 1369 | жен | 63 | 301 | 1.90 | |
| 1134 | муж | 32 | 53 | 1.70 | 1277 | жен | 64 | 330 | 1.70 | |
| 571 | муж | 33 | 30 | 1.80 | 1152 | жен | 65 | 112 | 1.70 | |
| 1161 | муж | 33 | 197 | 1.70 | 1205 | муж | 65 | 420 | 1.70 | |
| 1192 | жен | 33 | 110 | 1.70 | 237 | муж | 66 | 31 | 1.80 | |
| 1184 | жен | 34 | 305 | 1.70 | d559 | жен | 66 | 37 | 1.70 | |
| 1208 | муж | 35 | 207 | 1.60 | 1143 | жен | 67 | 84 | 1.60 | |
| 604 | жен | 36 | 52 | 1.90 | 1428 | муж | 67 | 433 | 1.80 | |
| 1086 | муж | 36 | 193 | 1.70 | 1365 | жен | 68 | 204 | 1.80 | |
| 1217 | муж | 36 | 407 | 1.60 | 173 | муж | 68 | 352 | 1.70 | |
| 1247 | жен | 36 | 286 | 1.70 | 1437 | муж | 68 | 213 | 1.80 | |
| 1235 | жен | 37 | 168 | 1.70 | 240 | муж | 69 | 18 | 1.90 | |
| 1345 | муж | 37 | 420 | 1.80 | 1146 | жен | 70 | 149 | 1.80 | |
| 1094 | жен | 38 | 172 | 1.70 | 1413 | муж | 70 | 522 | 1.90 | |
| 1219 | жен | 41 | 315 | 1.80 | 40 | жен | 72 | 179 | 1.80 | |
| 1381 | жен | 41 | 500 | 1.70 | 205 | жен | 72 | 461 | 1.80 | |
| 1207 | муж | 42 | 220 | 1.60 | 1115 | муж | 72 | 78 | 1.70 | |
| 1323 | муж | 42 | 300 | 1.80 | 1576 | муж | 72 | 307 | 1.80 | |
| 1411 | муж | 42 | 569 | 1.80 | 178 | муж | 73 | 182 | 1.70 | |
| 1145 | жен | 45 | 323 | 1.60 | 1669 | муж | 74 | 351 | 1.70 | |
| 1171 | жен | 45 | 67 | 1.60 | 121 | жен | 75 | 163 | 1.70 | |
| 1491 | жен | 46 | 498 | 1.70 | 1168 | жен | 79 | 239 | 1.70 | |
| 603 | жен | 47 | 108 | 1.90 | 1174 | жен | 79 | 271 | 1.60 | |
| 1155 | муж | 48 | 110 | 1.60 | 1160 | жен | 80 | 195 | 1.70 | |
| 1338 | муж | 49 | 696 | 1.80 | 55 | жен | 81 | 221 | 1.70 | |
| 1099 | муж | 50 | 144 | 1.70 | 56 | муж | 81 | 414 | 1.70 |
Как видно из таблицы, выделенные препараты ДНК имеют концентрацию в диапазоне от 30 нг/мкл до 784 нг/мкл и чистоту не ниже 1,6 по соотношению А260/А280.
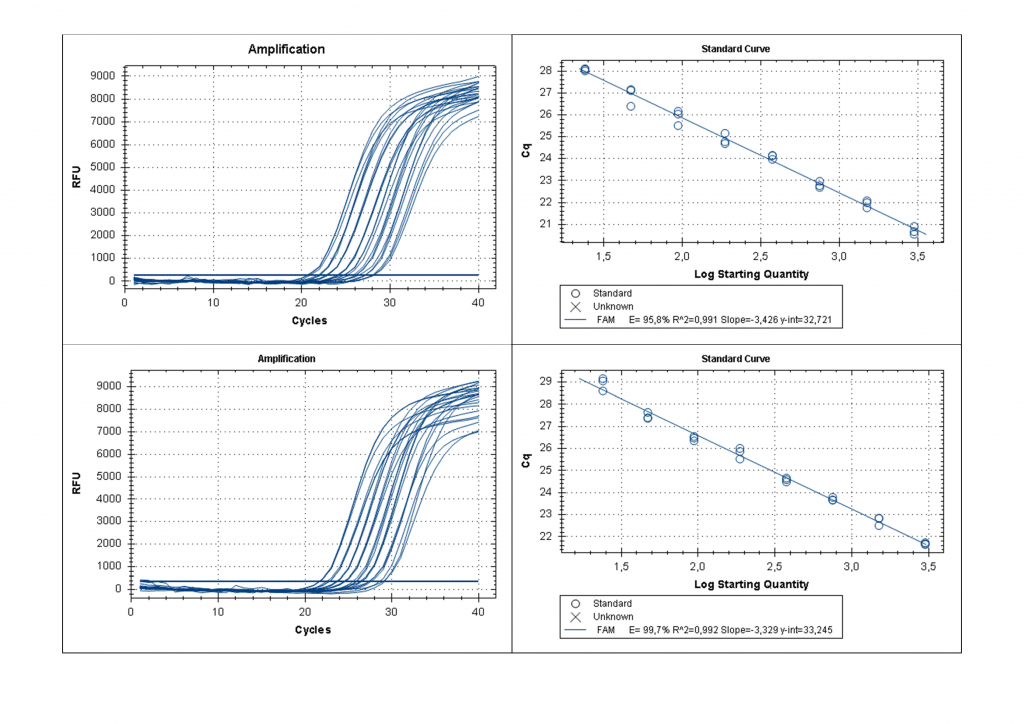
Полученные препараты ДНК расщеплялись одним из трех ферментов, и далее проводилась ПЦР, как указано в «Материалах и Методах». Гидролиз препаратов ДНК рестриктазой HinfI приводит к выщеплению анализируемого фрагмента, что позволяет избежать влияния остальной ДНК на результаты ПЦР. На рисунке 2 представлены кривые флуоресценции ПЦР для 8-ми разведений препаратов ДНК донора №1087 и ДНК клеточной линии Raji, гидролизованных ферментом HinfI, в диапазоне от 3000 до 24 копий.
Как видно из рис.2 и в соответствии с теорией ПЦР [9], при уменьшении количества ДНК-матрицы в 2 раза происходит увеличение Cq на 1 цикл и этот эффект наблюдается на всем диапазоне от 24 до 3000 копий ДНК в пробе. Таким образом, в результате расщепления анализируемого участка ДНК ферментами (при наличии соответствующих сайтов узнавания) происходит уменьшение количества молекул копируемой ДНК-матрицы и, соответственно, должно происходить увеличение величины Cq, как описано выше. В таблице 2 для каждого образца ДНК представлены значения Cq, полученные в ПЦР после расщепления ДНК ферментом HinfI (столбик 2), а также в ходе FatI- и GlaI-ПЦР анализа (столбики 3 и 4, соответственно). В столиках 5 и 6 указаны значения величин Cq, полученные в FatI- и GlaI-ПЦР анализе, за вычетом значения Cq в столбике 2, обозначенные как ΔCq_T и ΔCq_5mC, соответственно.
Таблица 2.
Значения Cq в случае гидролиза ДНК по сайтам GANTC (HinfI), CATG (Fat-ПЦР анализ) и A(5mC)GC (GlaI-ПЦР анализ) и значения разницы величины Cq для двух последних реакций относительно первой (ΔCq_T и ΔCq_5mC), соответственно.
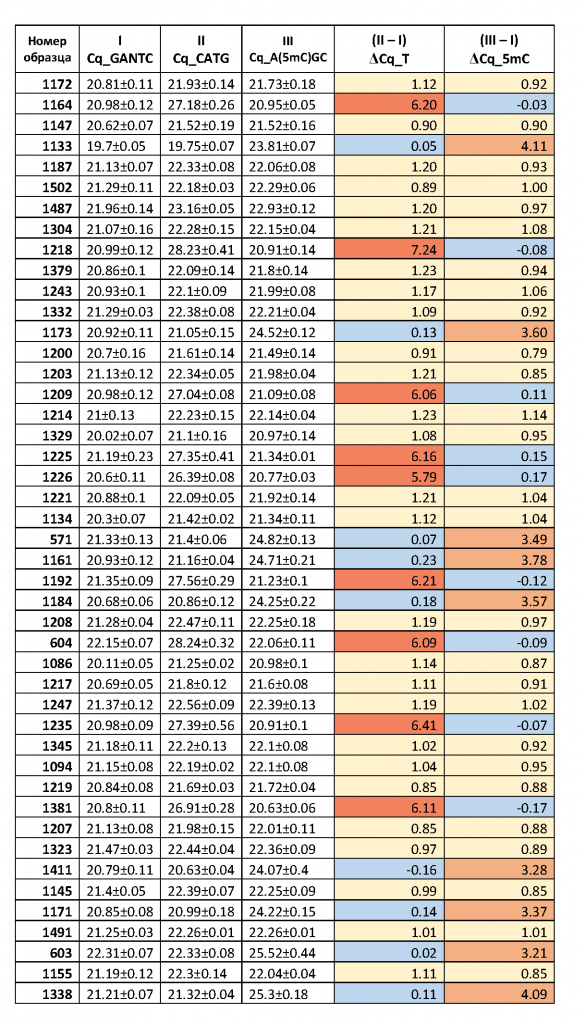
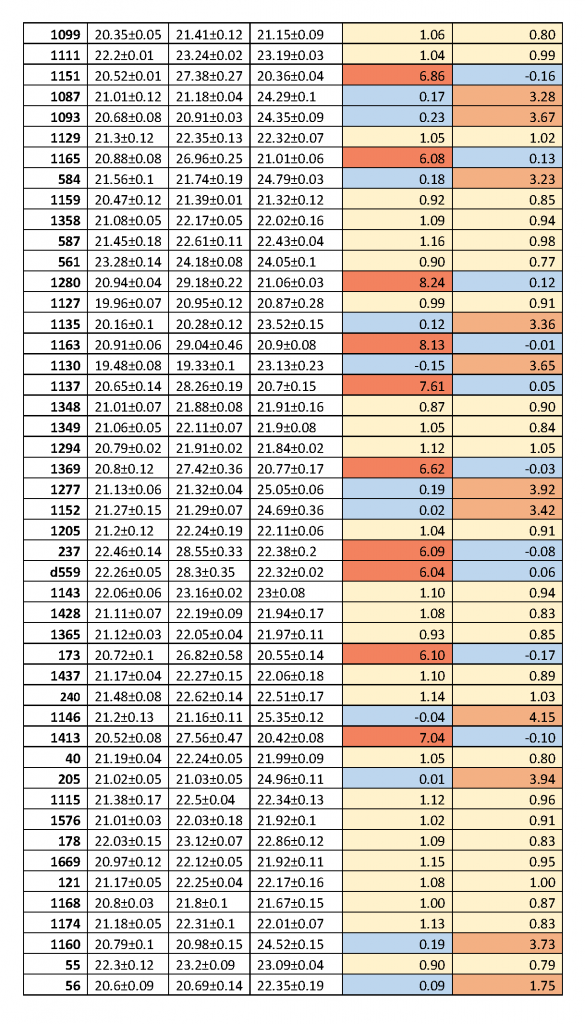
Как видно из таблицы 2 значения ΔCq_T, также как и ΔCq_5mC, отчетливо разбиваются на три группы величин. В первой группе значения ΔCq_T и ΔCq_5mC равны 0±0,23 и они отмечены голубым цветом. Во второй группе значения ΔCq_T и ΔCq_5mC варьируются в диапазоне 1±0,23 и они помечены желтым цветом. Третья группа значений ΔCq_T, выделенная красным цветом, находится в интервале значений 5,79-8,24. Третья группа значений ΔCq_5mC, окрашенная в оранжевый цвет, варьируется от 3,21 до 4,15, за исключением препарата ДНК за номером 56.
При этом, для всех образцов ДНК наблюдается соответствие значений ΔCq_T и ΔCq_5mC, представленное в трех вариантах. Нулевое значение ΔCq_T наблюдается при значениях ΔCq_5mC от 3,21 до 4,15 (1,75 для препарата ДНК за номером 56) – вариант 1. Нулевое значение ΔCq_5mC соответствует 3-ей группы значений ΔCq_T (5,79-8,24) – вариант 2. Наконец, вторая группа значений ΔCq (1±0,23) наблюдается только для ΔCq_T и ΔCq_5mC одновременно – вариант 3.
Первый вариант показывает значительное преобладание в анализируемой ДНК (обе хромосомы диплоидного набора) метилированного сайта A(5mC)GC, что соответствует гомозиготе по основанию С в позиции хр16: 75033884.
Во второй варианте высокие значения ΔCq_T свидетельствуют о том, что ДНК этих доноров гомозиготна по основанию T в позиции хр16: 75033884. В этом случае нет метилированного цитозина в этой позиции и, соответственно, нет сайта A(5mC)GC в положении хр16: 75033883. В итоге мы наблюдаем нулевые значения ΔCq_5mC.
В третьем варианте значения ΔCq_5mC и ΔCq_T близки к 1, что соответствует гетерозиготности ДНК доноров по позиции хр16: 75033884. В этом случае как в GlaI-ПЦР анализе, так и в FatI-ПЦР анализе расщепляется лишь одна из пары хромосом №16. При этом в обоих гидролизатах происходит уменьшение количества доступного для амплификации субстрата в 2 раза, что приводит увеличению значения Cq на один цикл.
Обращает на себя внимание образец ДНК 56, который имеет более низкое значение ΔCq_5mC по сравнению с остальными величинами в первой группе. Поскольку Fat-ПЦР анализ показывает отсутствие основания T в позиции хр16: 75033884, то, по-видимому, находящийся в этом положении цитозин является частично неметилированным. Однако, в связи с большим возрастом данного донора (81 год), небольшое значение ΔCq_5mC может быть связано с появлением в части клеток крови мутаций в ДНК, и в частности, в сайте A(5mC)GC, что будет приводить к отсутствию расщепления ДНК как в FatI-, так и в GlaI-ПЦР анализе.
Значения ΔCq_T от 5,79 и выше соответствуют гидролизу ДНК ферментом FatI на более чем 98%, что означает практически полный гидролиз ДНК. Ранее в GlaI-ПЦР анализе мы наблюдали гидролиз лейкоцитарной ДНК также на 98% [5]. Однако, при GlaI-ПЦР анализе фрагмента ДНК хр16: 75033860 – 75033957 мы получили значения ΔCq_5mC от 3,21 до 4,15, что означает гидролиз ДНК по сайту A(5mC)GC в положении хр16: 75033883 на глубину от 90 до 96%. Таким образом, не более 10% ДНК лейкоцитов крови имеют, видимо, неметилированный цитозин в положении хр16: 75033884.
На рисунке 3А результаты, представленные в таблице 2, отсортированы в порядке уменьшения ΔCq_5mC, а на рисунке 3Б показана доля молекул ДНК, нерасщепленных в ходе GlaI- и FatI-ПЦР анализа, относительно общего количество молекул ДНК, определенных ПЦР анализом HinfI-гидролизатов (выражена в процентах). Как видно из рисунка 3Б, суммарная доля молекул ДНК, нерасщепленных в ходе GlaI- и FatI-ПЦР анализа, близка к 100%, при этом на рисунках 3А и 3Б хорошо видны три варианта соответствия значений ΔCq_T и ΔCq_5mC, описанные выше.
Рисунок 3
Сверху – Значения ΔCq_5mC и ΔCq_T Cq для 92-х образцов донорской ДНК лейкоцитов (красным и синим цветом, соответственно). Снизу — процент молекул ДНК, нерасщепленных в ходе GlaI- и FatI-ПЦР анализа, относительно общего количество молекул ДНК, определенных ПЦР анализом HinfI-гидролизатов (красным и синим цветом, соответственно).
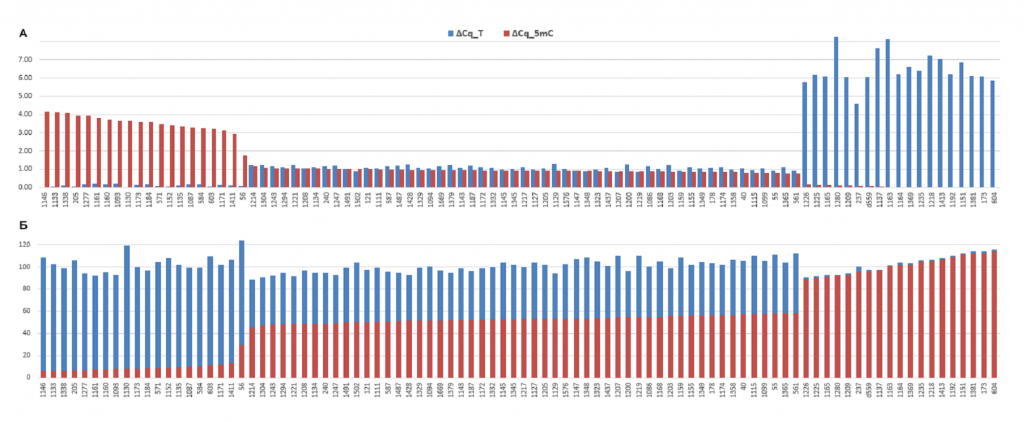
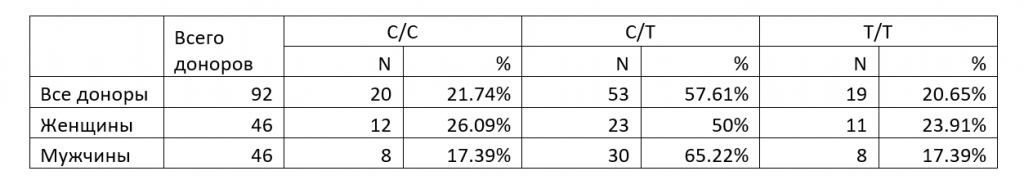
Из данных таблицы 2 следует, что 53 донора (57,61%) имеют гетерозиготный набор C/T в положении хр16: 75033884, 19 доноров (20,65%) гомозиготны по T, а 20 доноров (21,74%) – по C. Таким образом, принимая во внимание, что клетки крови имеют диплоидный набор хромосом, замена C на T встречается в 91 из 184 проанализированных вариантов (49,46%). Данная частота аллеля T в ДНК доноров несколько отличается от величины 56%, полученной ранее в исследовании геномной ДНК сибиряков [10], что, вероятно, связано с небольшой выборкой препаратов ДНК (28 образцов, из которых лишь 17 использовано для определения частот аллелей в рассматриваемой позиции), проанализированных в работе по секвенированию ДНК людей, живущих в Сибири.
В Таблице 3 показано число диплоидных сочетаний аллелей и их доля от общего количества проанализированных препаратов ДНК доноров крови, а также эти параметры отдельно для препаратов ДНК крови мужчин и женщин. Анализировались три возможных диплоидных сочетаний аллелей: гомозиготы по аллелям C/C и T/T и гетерозигота C/T. Интересно отметить, что для подвыборки женщин наблюдается соответствие частот генотипов, близкое к закону Харди-Вайнберга [11] (частоты 25-50-25% для частоты аллеля 0.5), в то время как у мужчин наблюдается существенное преобладание гетерозигот (65,22%).
Таблица 3
Распределение диплоидных наборов алеллей в общей выборке и отдельно по полам.
Дополнительно, по той же схеме были определены значения ΔCq_5mC и ΔCq_T для геномных ДНК, выделенных из нескольких клеточных линий (Таблица 4).
Таблица 4
Значения ΔCq для ряда геномных ДНК малигнантных клеточных линий человека и клеточной линии фибробластов легких L68
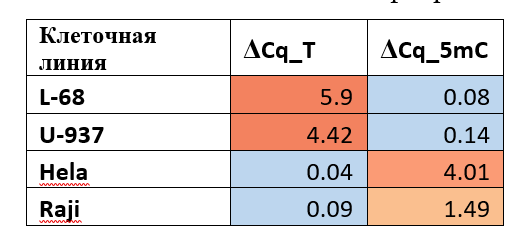
Результаты GlaI- и Fat-ПЦР анализа показывают, что геномы клеток L-68 и U-937 имеют в положении хр16: 75033884 набор T/T, в то время как в геноме клеток HeLa и Raji в данной позиции присутствует набор C/C в метилированной форме. При этом, однако, в клетках HeLa метилирование цитозина соответствует уровню метилирования в препаратах ДНК лейкоцитов большинства доноров, тогда как в клетках Raji уровень метилирования этого цитозина существенно ниже и близок к уровню метилирования препарата ДНК донора 56.
Таким образом, проведенные GlaI- и FatI-ПЦР анализы позволили определить число гомо- и гетерозиготных образцов ДНК в геномной позиции хр16: 75033884. Показано существенное преобладание метилированной формы цитозина в данном положении в препаратах ДНК лейкоцитов доноров. Факт преобладания 5mC позволяет определить число гомо- и гетерозиготных образцов ДНК методами GlaI- и FatI-ПЦР анализа независимо. Сравнение результатов GlaI- и FatI-ПЦР анализа показало их полное соответствие друг другу и подтвердило, таким образом, высокую эффективность предлагаемых методов GlaI- и FatI-ПЦР анализа. Оба метода, совместно или порознь, могут быть использованы для выявления SNP и в эпигенетических исследованиях для определения метилирования цитозиновых оснований в клинически значимых вариабельных участках генома.
Литература
- Youk J, An Y, Park S, Lee JK, Ju YS. The genome-wide landscape of C:G > T:A polymorphism at the CpG contexts in the human population. BMC Genomics. 2020; 21(1):270.
- Sherry ST, Ward MH, Kholodov M, Baker J, Phan L, Smigielski EM, Sirotkin K. dbSNP: the NCBI database of genetic variation. Nucleic Acids Res. 2001; 29(1):308-311.
- Tarasova, G.V., Nayakshina, T.N., Degtyarev, S.K. Substrate specificity of new methyl-directed DNA endonuclease GlaI. BMC Molecular Biol 9, 7 (2008).
- Jordà M, Díez-Villanueva A, Mallona I, et al. The epigenetic landscape of Alu repeats delineates the structural and functional genomic architecture of colon cancer cells. Genome Res. 2017; 27(1):118-132.
- EV Dubinin, AG Akishev, MA Abdurashitov, SB Oleynikova, VL Sitko, and S Kh Degtyarev Real time GlaI-PCR assay of regulation regions of human genes HDAC4, RARB and URB1 // Research Journal of Pharmaceutical, Biological and Chemical Sciences, vol 7(2), pp. 667-676 (2016).
- А.Г. Акишев, Н.А. Нетесова, М.А. Абдурашитов, И.В. Вихлянов, М.К. Никитин, А.Б. Карпов, С.Х. Дегтярев GlaI-ПЦР анализ метилирования сайта ACGC в хp11: 65647266 в препаратах ДНК клеток крови в норме и на ранних стадиях рака молочной железы // Эпигенетическая ДНК диагностика (2019).
- CFX96 and CFX384 Real-Time PCR Detection Systems. Instruction Manual. https://www.bio-rad.com/webroot/web/pdf/lsr/literature/10010424.pdf
- Инструкция по применению набора для очистки ДНК из лёгкой фракции клеток крови. http://russia.sibenzyme.com/info1561.php
- Kubista M, Andrade JM, Bengtsson M, Forootan A, Jonák J, Lind K, Sindelka R, Sjöback R, Sjögreen B, Strömbom L, Ståhlberg A, Zoric N. The real-time polymerase chain reaction. Mol Aspects Med. 2006 Apr-Jun;27(2-3):95-125.
- Whole-genome sequencing data of 28 modern-day humans from Siberia and Western Russia. https://www.ncbi.nlm.nih.gov/bioproject/PRJNA267856, https://www.ncbi.nlm.nih.gov/snp/rs66499664
- Stark AE, Seneta E. A reality check on Hardy-Weinberg. Twin Res Hum Genet. 2013 16(4):782-789.