Определение полиморфизма 5mC/T в позиции Chr1: 245618129 в препаратах ДНК из крови человека методами GlaI- и FatI-ПЦР анализа
А.Г. Акишев1 *, Н.А. Нетесова2, М.А. Абдурашитов1, С.Х. Дегтярев1
1 НПО «СибЭнзим» Новосибирск
2 ООО «Эпигенлаб» Новосибирск
* автор для переписки: Александр Акишев, НПО СибЭнзим, ул. Тимакова, д. 2/12 , Новосибирск, 630117, Россия; тел.: +7(383)333-4991; факс: +7(383)333-6853; E-mail: aki@sibenzyme.ru
Методами GlaI- и FatI-ПЦР анализа определяли частоты вариантов полиморфизма 5mC/T в положении хр1: 245618129 (по геномной сборке GRCh38.p13) в препаратах ДНК, выделенной из клеток крови 92 человек. Исследование включало (1) выделение лейкоцитарной ДНК из клеток крови, (2) проведение GlaI- и FatI-ПЦР анализа фрагмента ДНК хр1: 245617889 — 245618464, (3) определение 5-метилцитозина и тимина в позиции хр1: 245618129 в анализируемых препаратах ДНК и (4) сравнительный анализ полученных результатов.
Показано, что 43 донора (46,74%) имеют гетерозиготный набор C/T в положении хр1: 245618129, 28 доноров (30,43%) гомозиготны по T, а 21 донор (22,83%) – по C. Таким образом, принимая во внимание, что клетки крови имеют диплоидный набор хромосом, замена C на T встречается в 99 из 184 проанализированных вариантов (53,8%). При этом из полученных результатов следует, что цитозин в положении хр1: 245618129 в бОльшей части молекул ДНК находится в метилированной форме (5-метилцитозин).
Ключевые слова: однонуклеотидный полиморфизм 5mC/T, ДНК из крови человека, GlaI-ПЦР анализ, FatI-ПЦР анализ
DOI: 10.26213/SE.2019.69.42836
Данные для цитирования:
Акишев А.Г., Нетесова Н.А., Абдурашитов М.А., Дегтярев С.Х. (2021) Определение полиморфизма 5mC/T в позиции Chr1: 245618129 в препаратах ДНК из крови человека методами GlaI- и FatI-ПЦР анализа, Эпигенетич ДНК диагност, том 2021(1), с.13-23, DOI: 10.26213/SE.2019.69.42836

Эта статья доступна по лицензии Creative Commons Attribution-NonCommercial-NoDerivatives 4.0 International
Введение
Ранее нами было проведено изучение частот аллелей и статуса метилирования цитозинового остатка для полиморфизма C/T в положении хр16: 75033884 (по геномной сборке GRCh38) в препаратах ДНК 92 доноров [1]. В настоящей работе было проведено изучение другого однонуклеотидного полиморфизма C/T в положении хр1: 245618129 (rs7516354 по базе данных dbSNP [2]) в тех же препаратах ДНК. Данный SNP расположен в интроне гена KIF26B, кодирующего кинезин-подобный белок но, в отличие от SNP в позиции хр16: 75033884, не находится в ДНК-повторе.
На рисунке 1 представлена нуклеотидная последовательность участка геномной ДНК, включающая в себя анализируемый полиморфизм. Для проведения контрольной ПЦР в этом случае был подобран фермент рестрикции HaeIII, сайты узнавания которого расположены вне участка амплификации.
Рисунок 1. Нуклеотидная последовательность анализируемого участка геномной ДНК хр1: 245617889 — 245618464.
Фиолетовым фоном выделены сайты узнавания рестриктазы HaeIII (GGCC), окаймляющие амплифицируемый участок. Зоны связывания прямого и обратного праймеров, а также флуоресцентно-меченого TaqMan-зонда выделены желтым, голубым и зеленым фоном, соответственно. Подчеркнут сайт ACGC, который при метилировании расщепляется метилзависимой эндонуклеазой GlaI [3]. Цитозиновый остаток в этом сайте, который может замещаться тиминовым, выделен красным цветом.
При замене первого нуклеотида C в сайте ACGC на T возникает сайт узнавания рестриктазы FatI (CATG). Как было показано ранее [1], наличие SNP в сайте CACGC и определение замены 5mC на T в одной или обеих копиях хромосомы 1 диплоидного набора генома может быть установлено путем проведения Fat- и GlaI-ПЦР анализа и определения ΔCq_T и ΔCq_5mC при сравнении с данными контрольного ПЦР. При наличии неметилированного цитозина в позиции SNP в меньшей части молекул ДНК, GlaI-ПЦР анализ позволяет оценить долю 5mC.
Таким образом, методами GlaI- и FatI-ПЦР анализа мы определяли частоты вариантов полиморфизма 5mC/T в положении хр1: 245618129 (по геномной сборке GRCh38.p13) в препаратах ДНК, выделенной из клеток крови 92 человек.
Материалы и методы
FatI- и GlaI-ПЦР анализ образцов ДНК 92-х условно-здоровых доноров, выделенной из лейкоцитов периферической крови, проводили как описано ранее [1], за исключением замены эндонуклеазы рестрикции для контрольной ПЦР. Для выщепления амплифицируемого фрагмента при проведения контрольной ПЦР использовали рестриктазу HaeIII вместо фермента HinfI [1]. Возраст и пол доноров, а также концентрация полученных препаратов ДНК и их спектрофотометрическая чистота представлены в таблице 1 в работе [1].
Ниже указаны структуры праймеров (1-129d и 1-129r) и флуоресцентно-меченого зонда (1-129z), рассчитанных для проведения ПЦР-анализа участка хр1: 245618040 — 245618159:
1-129z 5′ FAM-CCTTTTCCCTCCCTCCCTGCCTCC-BHQ1 3′(24)
1-129d 5′ CCAAAGTCAGGGTTACAGAAGGAAGAGA 3′(28)
1-129r 5′ CTT TGT TGA GGA GGC GAT CGG AG 3′(23)
ПЦР в реальном времени проводили согласно протоколу производителя [4] в объеме 20 мкл на детектирующем амплификаторе CXF-96 («Bio-Rad», США) по следующей программе:
при 95°C — 3 мин; 5 «слепых» циклов: 95°C — 10 сек, 66°C — 20 сек, 72°C — 15 сек; 40 циклов: 95°C — 10 сек, 66°C — 20 сек (с детекцией флуоресцентного сигнала в канале FAM), 72°C — 15 сек.
По завершении ПЦР, при помощи программного обеспечения амплификатора “Bio-Rad CFX Manager v.2.1”, устанавливали среднее значение Cq и значение среднеквадратичного отклонения для анализируемых образцов N1, N2 и N3 как описано ранее [1] и полученные данные заносили в таблицу.
Результаты и обсуждение
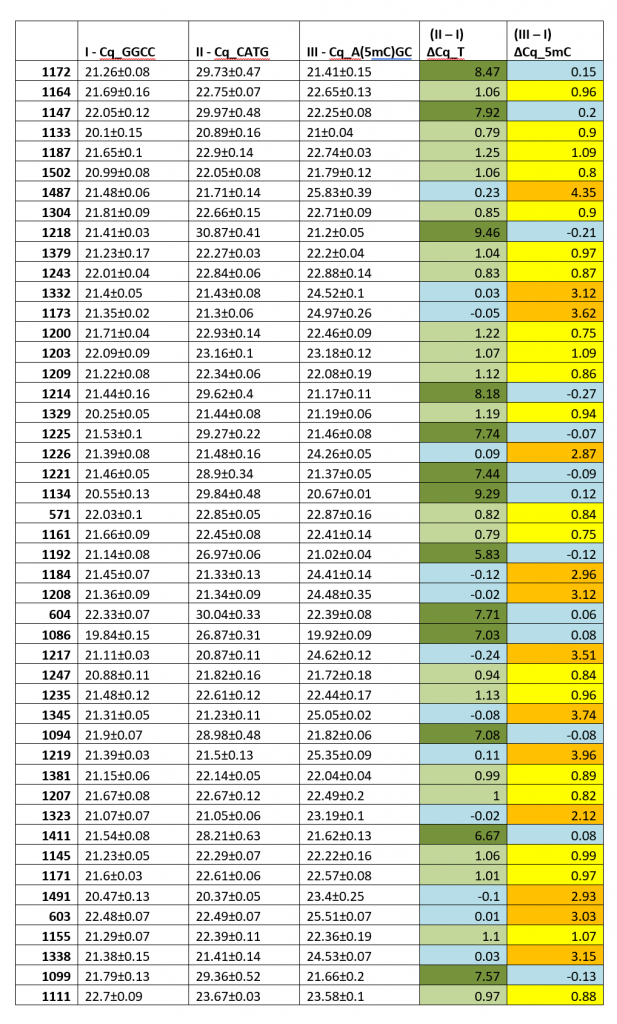
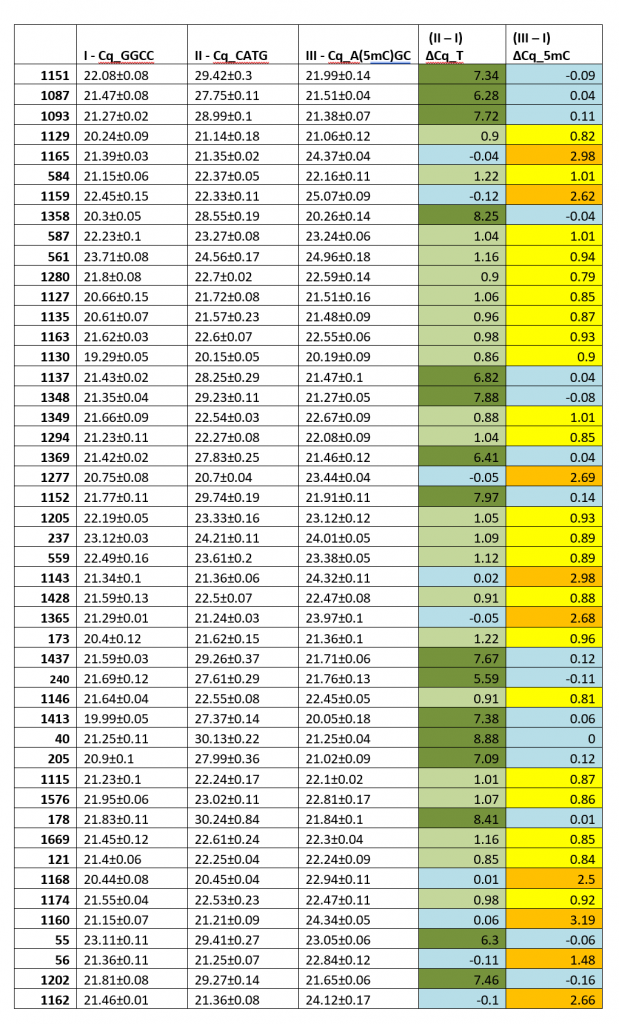
В Таблице 1 показаны значения Cq, полученные при ПЦР-анализе гидролизатов ДНК 92-х доноров крови (столбцы 2-4). В столбцах 5 и 6 указаны значения величин Cq, полученные в FatI- и GlaI-ПЦР анализе, за вычетом значения Cq в столбике 2 (контрольный ПЦР на HaeIII-гидролизате), обозначенные как ΔCq_T и ΔCq_5mC, соответственно.
Анализ данных таблицы 1 показывает, что как и в предыдущей работе [1], выявляются 3 группы образцов ДНК. В первой из них значения ΔCq_T и ΔCq_5mC варьируют от 0.75 до 1.25, что соответствует двукратному уменьшению количества нерасщепленных копий амплифицируемого участка. Таким образом, образцы первой группы представляют собой гетерозиготы по аллелям T:5mC в позиции хр1: 245618129.
Во второй группе значения ΔCq_T варьируют от 5.59 до 9.46, в то время как значения ΔCq_5mC минимальны (от -0.27 до 0.2). Очевидно, что образцы ДНК второй группы практически полностью расщепляются ферментом FatI и не содержат сайтов узнавания GlaI. Это говорит о том, что в исследуемой позиции SNP находится лишь тиминовый остаток (гомозиготы по T).
Для третьей группы образцов ДНК характерно повышенное значение ΔCq_5mC (2.12-4.35, за исключением образца №56) при небольших значениях ΔCq_T (от -0.24 до 0.23). Соответственно, данные образцы можно расценивать как содержащие лишь цитозиновый остаток в позиции хр1: 245618129 (гомозиготы по C), причем этот остаток находится преимущественно в метилированной форме.
Как и в работе [1], значения ΔCq_5mC (от 2.12 до 4.35) существенно меньше величин ΔCq_T (от 5.59 до 9.46), что также, вероятно, связано с наличием неметилированного цитозина, доля которого может достигать почти четверти. Кроме того, также как в работе [1], образец ДНК №56, имеет еще более низкое значение ΔCq_5mC по сравнению с остальными величинами в первой группе. Поскольку Fat-ПЦР анализ образца ДНК №56 показывает отсутствие основания T в позиции хр1: 245618129, то, по-видимому, доля неметилированного цитозина в этом положении свыше 30%.
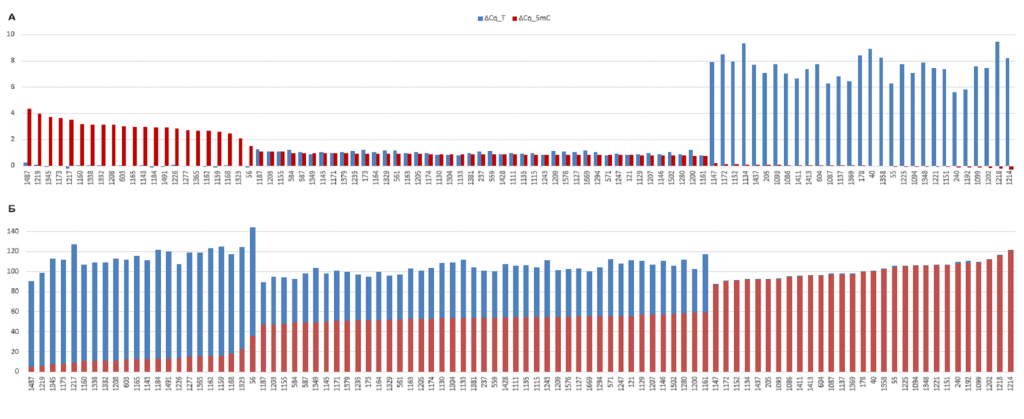
На Рисунке 2А результаты, представленные в таблице 1, отсортированы в порядке уменьшения ΔCq_5mC, а на рисунке 2Б показана доля молекул ДНК, нерасщепленных в ходе GlaI- и FatI-ПЦР анализа, относительно общего количество молекул ДНК, определенных ПЦР анализом HaeIII-гидролизатов (выражена в процентах). Как видно из рисунка 2Б, суммарная доля молекул ДНК, нерасщепленных в ходе GlaI- и FatI-ПЦР анализа, близка к 100%, при этом на рисунках 2А и 2Б хорошо видны три варианта соответствия значений ΔCq_T и ΔCq_5mC, описанные выше.
Рисунок 2.
Сверху – Значения ΔCq_5mC и ΔCq_T Cq для 92-х образцов донорской ДНК лейкоцитов (красным и синим цветом, соответственно). Снизу — процент молекул ДНК, нерасщепленных в ходе GlaI- и FatI-ПЦР анализа, относительно общего количество молекул ДНК, определенных ПЦР анализом HinfI-гидролизатов (красным и синим цветом, соответственно).
Таким образом, гетерозиготный набор С:Т имеется в 43 образцах ДНК, 28 образцов представляют собой гомозиготу T:T, а 21 образец является гомозиготным по аллелю C. Частота аллеля Т в исследованной выборке составляет 53.8%. Полученные данные показывают, что во всех образцах ДНК цитозиновый остаток преимущественно метилирован. Как было показано ранее, метилированная форма CG-динуклеотидов преобладает во всех компартментах генома за исключением CpG-островков и первых экзонов, выполняющих функцию регуляции экспрессии генов [5]. Нуклеотид в положении хр1: 245618129 расположен в шестом интроне гена KIF26B и также является в основном 5-метилцитозином.
В Таблице 2 показано число диплоидных сочетаний аллелей и их доля от общего количества проанализированных препаратов ДНК доноров крови, а также эти параметры отдельно для препаратов ДНК крови мужчин и женщин. Анализировались три возможных диплоидных сочетаний аллелей: гомозиготы по аллелям C/C и T/T и гетерозигота C/T для положения хр1: 245618129.
Таблица 2.
Распределение диплоидных наборов алеллей в позиции хр1: 245618129 в общей выборке и отдельно по полам.
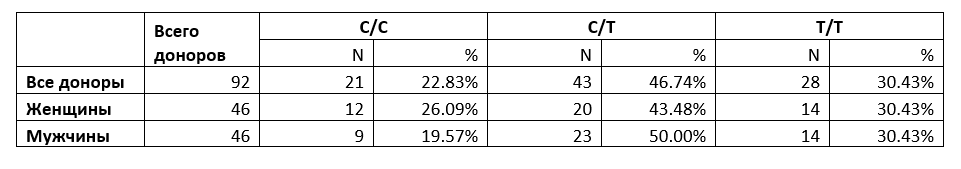
Как видно из таблицы 2, общее количество гетерозигот во всей выборке (46,74%) близко к значению 50%, которое и должно быть для такого распределения.
Ранее мы проводили определение SNP 5mC/T в положении хр16: 75033884 в той же выборке препаратов ДНК 92-х доноров [1]. В Таблице 3 представлены данные по распределению гетеро- и гомозигот в положении хр16: 75033884 для каждого из трех аллельных вариантов хр1: 245618129.
Таблица 3
Распределение гомо- и гетерозигот по аллелям C и T для двух позиций SNP.
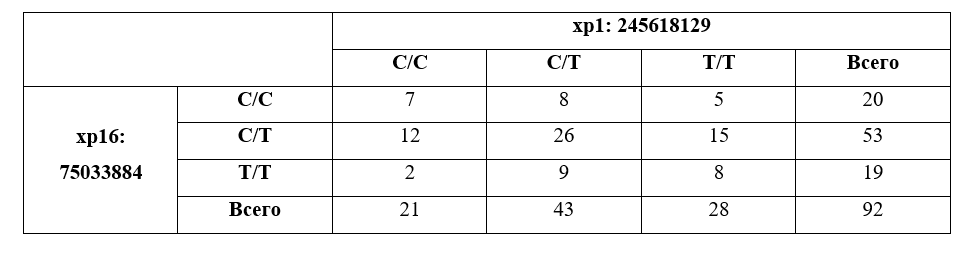
Как видно из таблицы, гетерозигота 5mC/T в положении хр16: 75033884 распределена приблизительно равномерно по все трем аллельным вариантам в положении хр1: 245618129. В тоже время комбинация гомозиготы по Т в положении хр16: 75033884 и гомозиготы по С в положении хр1: 245618129 встречаются существенно реже, чем другие аллельные варианты.
Мы также сравнили полученные количественные результаты с ожидаемыми по уравнению Харди-Вайнберга [6] для двух вариабельных позиций, исходя из полученных частот аллелей T=0.538 для хр1: 245618129 и T=0.495 для хр16: 75033884 (диаграмма на рисунке 3).
Рисунок 3
Количество различных генотипов по двум позициям SNP, полученных в эксперименте (столбики темно-синего цвета) и по расчетным данным (столбики светло-синего цвета). Заглавными буквами в подписях обозначены диплоидные варианты для позиции хр1: 245618129, прописными — для позиции хр16: 75033884.
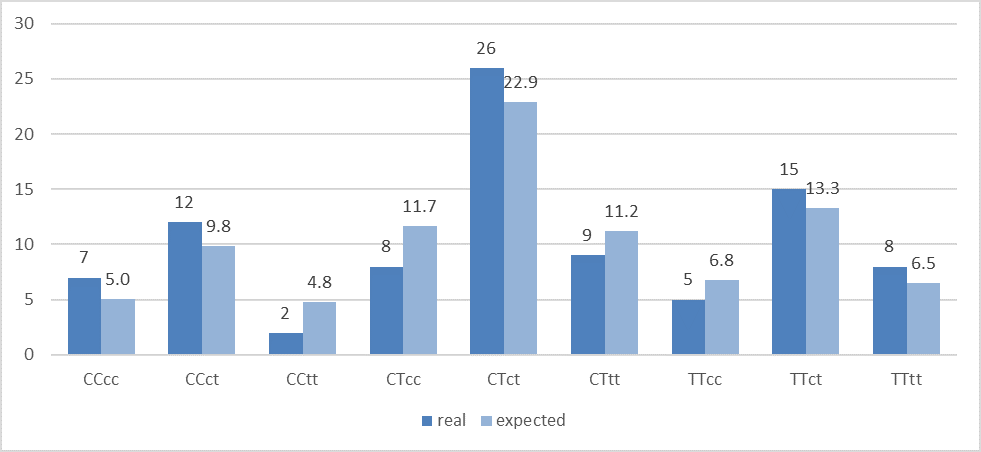
Как видно из рисунка 3, в целом, за исключением 3-ей пары столбиков, наблюдается соответствие экспериментальных результатов расчетным данным. Различие в паре столбиков CCtt, вероятно, связано с относительно небольшой изучаемой выборкой.
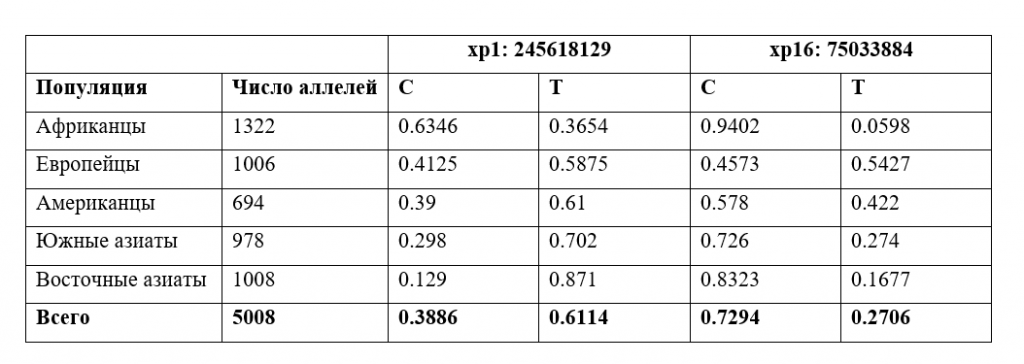
В таблице 4 приведены данные по частотам аллелей в позициях хр1: 245618129 и хр16: 75033884 для различных популяций по данным консорциума «1000 genomes» [7], из которого видно, что полученные нами данные наиболее соответствуют результатам SNP анализа ДНК представителей европейской популяции. Для европейцев установленные частоты аллелей T для позиций хр1: 245618129 и хр16: 75033884 составляют 0.588 [8] и 0.543 [9], соответственно, что близко к значениям частот 0.538 и 0.495 для этих позиций, полученных нами ранее [1] и в этой работе.
Таблица 4
Частоты аллелей C и T для двух позиций SNP по данным проекта «1000 геномов»
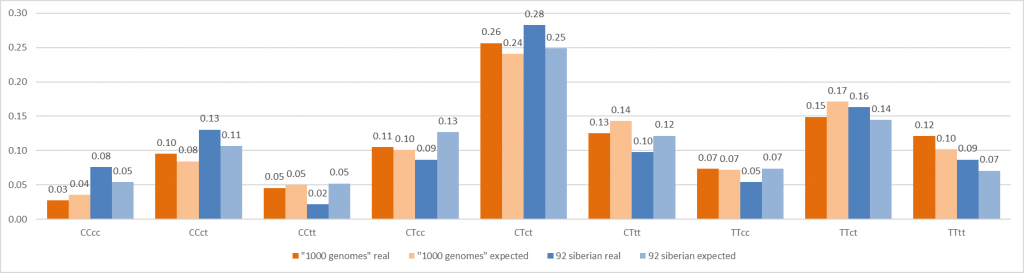
На рисунке 4 приведено сравнение экспериментальных данных, полученных при секвенировании 503 геномов (1006 аллелей) жителей Европы, с теоретическими расчетами в форме, аналогичной рис.3. Как видно из сравнения рисунков 3 и 4, картины распределения генотипов по позициям хр1: 245618129 и хр16: 75033884, выявленные у европейцев и полученные в нашей работе, отличаются незначительно. На рисунке 5 эти данные представлены вместе в виде сравнения по частотам SNP. Как видно из рис.5 кратные различия в частотах SNP для двух выборок, полученные в эксперименте, наблюдаются в случае гомозигот по С в обоих сайтах, а также в случае гомозиготы по С для позиции хр1: 245618129 и гомозиготы по Т для позиции хр16: 75033884. И если второй случай, как было отмечено выше, можно объяснить малым размером выборки в нашей работе, то различие в доле гомозигот по С в обоих положениях является, по-видимому, отражением реальных различий генотипов в изучаемых выборках ДНК жителей Сибири и Европы. Известно, что распределение гомо- и гетерозигот в реально существующих популяциях сильно зависит от миграционных потоков и выделения в обществе более или менее обособленных субпопуляций [10], что характерно в целом для современного мира.
Рисунок 4
Количество различных генотипов по двум позициям SNP, полученных в эксперименте (столбики темно-оранжевого цвета) и по расчетным данным (столбики светло-оранжевого цвета) для 503 геномов европейской популяции из базы данных «1000 геномов»
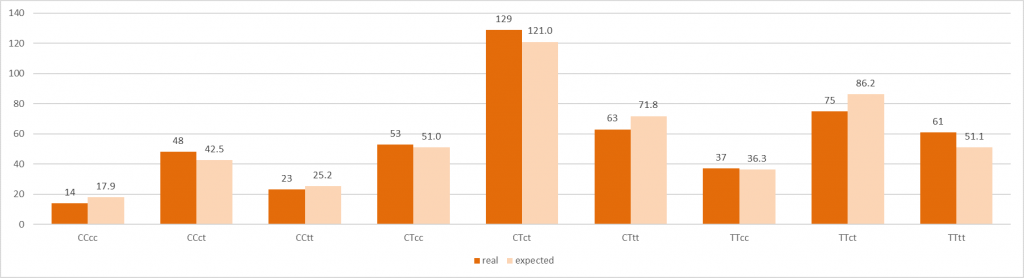
Рисунок 5
Сравнение частот различных генотипов по двум позициям SNP, полученных в настоящем исследовании с таковыми для 503 геномов европейской популяции из базы данных «1000 геномов»
Таким образом, с помощью методов FatI- и GlaI-ПЦР анализа нами были определены генотипы 92-х доноров по позиции хр1: 245618129, которая находится в интроне гена KIF26B, кодирующего кинезин-подобный белок. Показано, что 43 донора (46,74%) имеют гетерозиготный набор C/T в положении хр1: 245618129, 28 доноров (30,43%) гомозиготны по T, а 21 донор (22,83%) – по C. Принимая во внимание, что клетки крови имеют диплоидный набор хромосом, замена C на T встречается в 99 из 184 проанализированных вариантов (53,8%). При этом GlaI-ПЦР анализ показал, что цитозин в положении хр1: 245618129 в бОльшей части молекул ДНК находится в метилированной форме (5-метилцитозин), однако доля неметилированного цитозина может достигать 1/5 и более.
Методы FatI- и GlaI-ПЦР анализа могут быть использованы в медико-генетических исследованиях для установления предрасположенности к тем или иным заболеваниям и определения частоты нежелательных аллелей в данной географической местности или в относительно изолированных популяциях. Использование GlaI-ПЦР анализа позволяет также определить степень метилирования цитозина в случае, если он является одним из вариантов SNP. В настоящее время установлена связь между метилированием определенных участков генома и развитием онкологических и других патологий, что делает актуальным внедрение подобных эпигенетических методов диагностики в практику [11].
Литература
- А.Г. Акишев, Н.А. Нетесова, М.А. Абдурашитов, Дегтярев (2021) Определение полиморфизма 5mC/T в повторе AluSx (CHR16: 75033884) в препаратах ДНК из крови человека методами GlaI- и FatI-ПЦР анализа. Эпигенетич ДНК диагност. том 2021(1), https://www.epigendx.online/ru/2021/snp/
- Sherry ST, Ward MH, Kholodov M, Baker J, Phan L, Smigielski EM, Sirotkin K. dbSNP: the NCBI database of genetic variation. Nucleic Acids Res. 2001;29(1):308-311.
- Tarasova, G.V., Nayakshina, T.N., Degtyarev, S.K. Substrate specificity of new methyl-directed DNA endonuclease GlaI. BMC Molecular Biol 9, 7 (2008).
- CFX96 and CFX384 Real-Time PCR Detection Systems. Instruction Manual. https://www.bio-rad.com/webroot/web/pdf/lsr/literature/10010424.pdf Accessed May 01 2021
- Edwards JR, Yarychkivska O, Boulard M, Bestor TH. DNA methylation and DNA methyltransferases. Epigenetics Chromatin. 2017;10:23.
- Stark AE, Seneta E. A reality check on Hardy-Weinberg. Twin Res Hum Genet. 2013 16(4):782-789.
- Sudmant, P., Rausch, T., Gardner, E. et al. An integrated map of structural variation in 2,504 human genomes. Nature 526, 75–81 (2015).
- dbSNP Short Genetic Variations. Reference SNP (rs7516354) Report https://www.ncbi.nlm.nih.gov/snp/rs7516354#frequency_tab Accessed May 01 2021
- dbSNP Short Genetic Variations. Reference SNP (rs66499664) Report https://www.ncbi.nlm.nih.gov/snp/rs66499664#frequency_tab Accessed May 01 2021
- Grover VK, Cole DE, Hamilton DC. Attributing Hardy-Weinberg disequilibrium to population stratification and genetic association in case-control studies. Ann Hum Genet. 2010;74(1):77-87.
- Chen Y, Hong T, Wang S, Mo J, Tian T, Zhou X. Epigenetic modification of nucleic acids: from basic studies to medical applications. Chem Soc Rev. 2017;46(10):2844-2872.